下一代测序(NGS)技术改变了临床研究人员和科学家对遗传学的看法,因为它在一次试验中评估多个基因。它可以在短时间内对感兴趣的整个或特定基因组进行测序。几种不同的NGS平台使用其他测序技术。不过,有一件事是共同的,即“所有的NGS平台都执行数百万个小片段的测序DNA并行执行。“然后使用生物信息学分析将这些片段拼凑在一起。
下一代测序原理
下一代测序技术与之类似毛细管电泳(CE)测序,其中DNA聚合酶催化荧光标记的脱氧核糖核苷酸三磷酸(dNTPs)合并到DNA模板链在连续的周期DNA合成.在每个过程中,通过添加每个核苷酸时的荧光团激发来识别核苷酸。主要的区别在于,NGS不是对单个DNA片段进行测序,而是同时将这一过程扩展到数百万个片段。NGS提供高精度,无错误读数的高产量,以及高于Q30的基础调用的高比例。
下一代测序的基本步骤
Illumina测序仪包括测序的四个基本步骤:文库准备、聚类生成、测序和分析。在分离出DNA或cDNA(从RNA合成)后,它们经历了以下四个基本步骤:
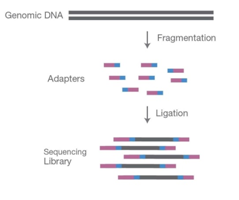
文库准备在测序过程中是至关重要的,因为它准备的样品与测序仪兼容。通过超声或酶切对DNA或cDNA进行裂解,得到长度为200-500 bp的片段。在碎片化过程中,每个片段在末端都有一个悬垂的A尾,准备将它们连接到适配器序列上,其中包含一个与A尾片段互补的' T '基悬垂。
适配器序列包含引物结合位点,索引序列,以及允许库片段附加到流单元草坪的序列。这些适配器连接在5 '和3 '端。另外,一种称为tagentation的过程将碎片和连接反应结合在一个步骤中,提高了文库制备过程的效率。
它有助于在PCR步骤中特异性富集适配器连接的DNA。

对于每个样品,都使用唯一的适配器,所有的样品都集中在一个管中。在池化后,将其加载到测序器中,以便进一步进行测序。
集群的一代
准备好的库装载在流单元中,并放置在序列器中。流池是一个玻璃载玻片,有1、2或8个物理分隔的通道,表面涂有一层表面绑定的、适配器互补的低聚物。目前,最常用的流池是采用半导体制造技术生产的模式化流池。它有一个玻璃衬底,含有带有DNA探针的图案化纳米孔,可以捕获制备好的DNA链,以便在集群生成期间进行扩增。

一旦样品附着在流池上并发生桥式扩增,簇的生成就开始了.在这个过程中,文库的DNA片段与流细胞表面的寡核苷酸之一杂交。然后,通过DNA聚合酶延长附着在流细胞上的低聚物来生成互补链。原来的分子被冲走,链像桥一样弯曲,附着在流池中的下一个低聚物上。这第二个寡核苷酸与另一个适配器序列互补,聚合酶生成互补链,形成双链桥。这个桥变性,导致两个分子的单链副本被拴在流细胞上。这个过程一遍又一遍地重复,同时发生数百万个簇,导致所有克隆扩增。
桥式扩增后,反向链被切割并洗掉,只留下正向链。3 '端被阻塞,以防止不必要的启动。当集群生成完成后,模板就可以进行排序了。

合成测序
流式细胞的每一个簇的测序都是一致的。测序所需的组分包括测序引物、DNA聚合酶和用荧光团荧光标记。但是,根据各自机器中使用的化学(4通道化学、2通道化学和1通道化学),要么所有核苷酸都被标记,要么只有少数核苷酸被荧光团标记
在四通道化学中,所有的核苷酸都用四种荧光染料标记。双通道化学使用了两种不同的荧光染料,而一个频道化学只使用一种染料。

在四通道化学的例子中,所有上述成分通过流池,在流池中,测序引物退火到适配器上的互补位置,DNA聚合酶添加用荧光团标记的互补核苷酸。这个荧光团作为一个阻断基团,DNA聚合酶不能添加另一个核苷酸,直到它被移除,允许探测器记录每个碱基被添加的荧光。加入每个核苷酸后,簇中的荧光团被光源激发,并记录发出的特征荧光信号。这个过程叫做sequencing-by-synthesis.

如何读取NGS中的核苷酸?
在测序过程中,在不同的点读取DNA片段的不同片段,一个簇包含多个读取。一旦流式细胞充满所有成分(DNA聚合酶、测序引物和荧光标记的核苷酸),测序引物退火到基因组DNA的左侧,从5 '侧读取片段并考虑先读或先读1.然后,这些成分被洗掉,模板的3 '端被去保护,这使得它可以折叠并结合到流单元上的低聚物上。
流池再次充满所有成分,但添加了与正确适配器互补的引物。这将解码在合适的适配器中找到的索引,并将其视为二读或二读。在每个周期后,荧光团发射检测每个集群使用光学系统,并确定基础纳入。这个从两端确定顺序的过程叫做paired-end测序。与此同时,在single-read测序,序列只从库片段的一端确定。
数据分析
测序完成后,光信号被翻译成核苷酸序列称为基本要求.基调用的准确性由Phred质量评分(问分数),这是评估测序数据质量的最常用指标。Q分数表示给定碱基被排序器错误调用的概率。
Q分数被定义为与基本调用错误概率(P)对数相关的属性2.Q= - 10log10P
这个Q值决定了一个好的或坏的基础。质量评分和基本呼叫精度如下所示。评分Q30是一系列测序应用的理想选择。
在图书馆筹建期间,每个样本被赋予唯一的索引序列,这被称为多路复用;这使得大量的库可以合并在一起,并在一次测序运行中同时进行测序。所以,在数据分析之前,有一个过程叫做多路分解发生,它根据序列的惟一索引将其从池中的示例库中分离出来。对于每个样本,具有相似碱基延伸的读取都是局部聚类的。正向和反向读取是成对的,创建连续的序列。这些新识别的连续序列与参考序列对齐。

与参考基因组相匹配的读取数是至关重要的深度报道.覆盖深度越高,越多的数字具有相同的序列和对齐。这种比对显示了参考基因组和可以使用生物信息学工具识别的分离样本序列之间的差异或相似之处。
在校准后,许多分析变化是可能的,如单核苷酸多态性(SNP)或插入-缺失(indel)鉴定,RNA方法的读取计数,系统发育或宏基因组分析,等等。
新一代测序技术的应用
下一代测序技术应用广泛。以下是其中一些:
- NGS可用于进行宏基因组测序(宏基因组测序是通过对不同生物体使用多组引物从环境或临床样本中识别生物体的过程),用于检测未知疾病相关病毒和新型人类病毒。
- 在人类基因组中,只有不到2%的基因组由包含大多数已知致病变异的外显子组组成,使用NGS进行全外显子组测序具有成本效益。
- 这种方法不仅用于人类基因组测序,还用于其他物种测序,如农业上重要的牲畜、植物或与疾病相关的微生物。
- 全基因组测序分析整个基因组并产生大量数据,使其成为基因组学研究的有力工具。不同的测序仪在短时间内对样品进行测序,如下所述:
下一代测序的局限性
NGS有一些限制,如下所述:
- 它需要复杂的生物信息学系统、快速数据处理和大数据存储能力,因此成本很高。
- 测序前PCR扩增可能导致文库准备过程中的PCR偏差(序列gc含量、片段长度和假多样性)和分析(基础误差/偏向某些序列)。
参考文献
- 陈志强,陈志强,陈志强。金宝搏beat亚洲体育真人荷官分子生物学。2019。240 - 269 p。
- https://www.illumina.com/science/technology/next-generation-sequencing/beginners/ngs-workflow.html
- https://medium.com/@tiffanysouterre/dna-sequencing-techniques-explained-53c21eef51b1
- https://www.cd-genomics.com/blog/principle-and-workflow-of-illumina-next-generation-sequencing/
- Illumina公司网站
- https://www.technologynetworks.com/genomics/articles/an-overview-of-next-generation-sequencing




